Désorption-ionisation laser assistée par matrice
La désorption-ionisation laser assistée par matrice est une technique d'ionisation douce utilisée en spectrométrie de masse, donnant la possibilité l'analyse de biomolécules...
Recherche sur Google Images :
Source image : www.univ-lille1.fr Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Page(s) en rapport avec ce sujet :
- Ce mémoire sur les mécanismes de la désorption / ionisation laser assistée par matrice (MALDI) est le fruit de travaux réalisés au Laboratoire de ... (source : univ-lille1)
- ... La désorption - ionisation laser assistée par matrice (en anglais Matrix.... avec la MALDI est le TOF (spectromètre de masse à temps de vol... (source : cat.inist)
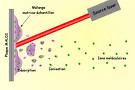
La désorption-ionisation laser assistée par matrice (en anglais Matrix Assisted Laser Desorption Ionisation ou MALDI) est une technique d'ionisation douce utilisée en spectrométrie de masse, donnant la possibilité l'analyse de biomolécules (des biopolymères comme les protéines, les peptides et les sucres) et les grosses molécules organiques (comme les polymères, les dendrimères et autres macromolécules) qui tendent à devenir fragiles ainsi qu'à se fragmenter quand elles sont ionisées par des méthodes plus conventionnelles. Elle est assez comparable à l'ionisation par électrospray en douceur relative et en ions produits (quoiqu'elle crée moins d'ions multichargés).
L'ionisation est génèrée par un faisceau laser (normalement un laser à l'azote). Une matrice est utilisée pour protéger la biomolécule de la destruction par un faisceau direct et de favoriser la vaporisation et l'ionisation.
Matrice
La matrice consiste en des molécules cristallisées, qui sont généralement choisies parmi les trois suivantes : l'acide 3, 5-diméthoxy-4-hydroxycinnamique (acide sinapinique), l'acide α-cyano-4-hydroxycinnamique (alpha-cyano ou alpha-matrice) et l'acide 2, 5-dihydroxybenzoïque (DHB). Une solution d'un type de ses molécules est faite, quelquefois dans un mélange d'eau ultra-pure et de solvant organique (généralement de l'acétonitrile (ACN) ou de l'éthanol). L'acide trifluoroacétique (TFA) peut quelquefois être ajouté. Un bon exemple de solution-matrice est , par exemple, un mélange de 20 mg/ml d'acide sinapinnique dans l'ACN :eau :TFA (50 :50 :0, 1).
La nature des composants pour une matrice fiable est déterminée par tâtonnement avec essais et erreurs, mais elle est basée sur des considérations spécifiques de «profils moléculaires». Ses composants :
- sont de faible masse moléculaire (pour favoriser la vaporisation), mais assez grosses (avec une pression de vapeur assez grande) pour ne pas s'évaporer durant la préparation de l'échantillon ou lors de son introduction dans le spectromètre.
- sont acides, ou au moins agissant comme source de proton (s) pour faciliter l'ionisation de l'analyte.
- ont une forte absorption dans l'ultraviolet, leur permettant d'absorber efficacement et rapidement l'irradiation laser.
- sont fonctionnalisés avec des groupes polaires, pour une utilisation en solutions aqueuses.
La solution matrice est mélangée avec l'analyte (l'échantillon). Le solvant organique permet aux molécules hydrophobes de se dissoudre dans la solution, tandis que l'eau permet aux molécules hydrophiles de faire de même. La solution est ensuite déposée sur une coupelle MALDI (généralement en métal conçue pour cet usage). Le solvant se vaporise, laissant uniquement la matrice recristallisée, mais avec désormais des molécules d'analyte réparties dans tout le cristal. La matrice et l'analyte sont dits alors co-cristallisés dans un spot MALDI.
Liste de composés utilisables comme matrice
| Composé | Abréviation | Utilisation |
| Peptide (moins de 10kDa) Hydrate de carbone |
||
| Acide sinapinique | SA | Protéine (plus de 10kDa) Dendrimers Fullerenes |
| Protéine (plus de 10kDa) | ||
| Acide succinique | - | Peptide et protéine, Laser IR Polymère synthétique, Laser IR |
| - | Peptide et protéine, Laser UV | |
| - | Peptide et protéine, Laser UV | |
| - | Peptide et protéine, Laser UV | |
| Glycérol | - | Peptide et protéine, Matrice liquide |
| - | Peptide et protéine, Matrice liquide | |
| Oligonucléotide de moins de 3, 5kDa Hydrate de carbone acide |
||
| Oligonucléotide de plus de 3, 5kDa | ||
| Acide anthranilique | - | Oligonucléotide de plus de 3, 5kDa |
| Acide nicotinique | - | Oligonucléotide de plus de 3, 5kDa |
| IAA | Polymère synthétique non polaire | |
| DIT | Polymère synthétique non polaire Lipide Dendrimers |
|
| Polymère synthétique polaire Molécule organique Hydrate de carbone |
||
| - | Molécule organique | |
| - | Hydrate de carbone | |
| Molécule inorganique | ||
| - | Oligosaccharide | |
| - | Oligonucléotide | |
| Protéine (plus de 10kDa) |
Laser
Le laser est dirigé sur les cristaux du spot MALDI. Le spot absorbe l'énergie laser et il semble que la matrice est ionisée en premier par ce fait, puis transférerait une partie de sa charge aux molécules à étudier, les ionisant tout en les protégeant d'une énergie disruptive du laser. Les ions observés après cette étape sont quasi-moléculaires, ionisés par l'addition d'un proton en [M+H]+, ou d'un autre cation comme l'ion sodium [M+Na]+, ou par la soustraction d'un proton [M-H]- par exemple. La technique MALDI produit généralement des ions monochargés, mais des ions multichargés peuvent aussi être observés, selon la matrice utilisée et/ou l'intensité du laser. Ces espèces possèdent toutes un nombre pair d'électrons. Des signaux correspondants à des cations radicalaires peuvent aussi être observés, par exemple dans le cas des molécules de la matrice ou d'autres molécules stables.
MALDI à pression atmosphérique
L'AP-MALDI (Atmospheric pressure matrix-assisted laser desorption/ionization) est une technique d'ionisation (source d'ions) qui au contraire de la MALDI sous vide opère dans un environnement à pression atmosphérique[1]. La différence principale entre la MALDI sous vide et l'AP MALDI est la pression sous laquelle les ions sont créés. Dans la MALDI sous vide, les ions sont produits sous 10 mTorr ou moins tandis que les ions AP MALDI sont constitués sous pression atmosphérique. Le désavantage de la source AP MALDI est l'observation d'une sensibilité limitée mais aussi son intervalle de masse restreint.
La technique AP MALDI est utilisée en spectrométrie de masse pour des applications variant des champs protéomiques à la découverte de nouveaux médicaments. Les sujets les plus «populaires» auxquels s'adresse la technique sont : la protéomique, ADN/ARN/ANP, lipides, oligosaccharides, phosphopeptides, bactéries, petites molécules et polymères synthétiques. Des applications identiques sont disponibles pour la technique sous vide. La source AP MALDI peut être aisément couplée à un piège à ions de spectromètre de masse[2] ou tout autre dispositif de spectrométrie de masse équipé avec une source ESI (ionisation par électrospray) ou nanoESI.
Spectrométrie de masse (MALDI-TOF)
Le type de spectrométrie de masse généralement utilisé avec la MALDI est le TOF (spectromètre de masse à temps de vol - time-of-flight mass spectrometer), essentiellement à cause de sa large plage de fonctionnement en masse.
Historique
L'expression anglaise de matrix-assisted laser desorption ionization (MALDI) a été forgé en 1985 par Franz Hillenkamp, Michæl Karas et leurs collègues [3]. Ces chercheurs ont découvert que l'alanine, un acide aminé, pouvait être ionisé plus aisément si elle était mélangé avec le tryptophane (autre acide aminé) et irradié avec un laser pulsé à 266 nm. Le tryptophane absorbait l'énergie laser et aidait à ioniser l'alanine non absorbante. Les peptides jusqu'à la peptide 2843 Da (melittine) peuvent être ionisées quand elles sont mélangées avec ce type de «matrice»[4]. L'avancée pour la désorption-ionisation laser pour une grosse molécule se produisit en 1987 quand Koichi Tanaka (de Shimadzu Corp. ) et ses collègues utilisèrent ce qu'ils appelèrent la «méthode de la matrice métal ultra-fin/liquide» qui combinait des particules de 30 nm de cobalt dans du glycérol avec un laser à azote à 337 nm pour l'ionisation[5]. En utilisant la combinaison d'un laser et de la matrice, Tanaka était capable d'ioniser des biomolécules aussi importante que la protéine 34 472 Da carboxypeptidase-A. Tanaka reçut (conjointement) le prix Nobel de chimie 2002 pour avoir démontré, qu'avec une combinaison correcte de matrice et de laser, qu'une protéine pouvait être ionisée[6]. Karas et Hillenkamp furent ensuite capables d'ioniser l'albumine (protéine 67 kDa) en utilisant une matrice d'acide nicotinique et un laser à 266 nm[7]. Des améliorations supplémentaires furent réalisées en utilisant un laser à 355 nm et les dérivées de l'acide cinnamique, l'acide férulique, l'acide caféique et l'acide sinapinique comme matrice[8]. La disponibilité de lasers à azote fonctionnant à une longueur d'onde de 337 nm petits et assez peu coûteux et les premiers instruments commerciaux introduits au début des années 1990 conduit de nombreux chercheurs à utiliser la technique MALDI[9]. Actuellement, la spectrométrie de masse MALDI utilise essentiellement des matrices organiques.
Des macromolécules synthétiques, comme les caténanes et les rotaxanes, les dendrimères, les polymères hyper-ramifiés mais aussi d'autres ensembles moléculaires, ont des masses molaires de l'ordre de milliers ou de dizaines de milliers de grammes par mole, ce qui fait que la majorité des techniques d'ionisation rencontrent des difficultés à produire des ions moléculaires. La MALDI est une méthode analytique simple et rapide qui permet aux chimistes d'analyser les résultats de ce genre de synthèses et de vérifier leurs résultats.
En biochimie
En protéomique, la MALDI est utilisée pour l'identification des protéines isolées par électrophorèse sur gel : SDS-PAGE et électrophorèse sur gel à deux dimensions. Une méthode utilisée est l'empreinte de masse de peptide par MALDI-MS, ou par affaiblissement post-ionisation ou dissociation induite par collision (voir spectrométrie de masse).
Problèmes
La préparation d'échantillon pour MALDI est importante pour le résultat. Les sels inorganiques qui sont aussi des parties des extraits de protéines interfèrent avec le processus d'ionisation. Les sels sont retirés par une extraction en phase solide ou en lavant les spots cibles finaux à l'eau. Les deux méthodes peuvent retirer d'autres substances de l'échantillon. Le mélange de protéines de la matrice n'est pas homogène à cause de la différence de polarité qui conduit à la séparation des deux substances lors de la cristallisation. Le diamètre du spot de la cible est plus arge que celui du laser, ce qui rend indispensable de procéder à plusieurs tirs lasers en différents lieux de la cible, afin d'obtenir la moyenne statistique de la concentration de la substance dans le spot cible. La composition de la matrice, l'ajout d'acide trifluoroacétique et d'acide formique, le délai entre les impulsions, le temps d'attente pour la puissance d'accélération, la longueur d'onde du laser, la densité d'énergie du laser et l'angle d'impact du faisceau laser font partie des autres données critiques pour la qualité et la reproducibilité de la méthode.
Références
- ↑ (en) Laiko VV, Baldwin MA, Burlingame AL, «Atmospheric pressure matrix-assisted laser desorption/ionization mass spectrometry», dans Anal. Chem. , vol. 72, no 4, 2000, p. 652-7 [texte sur PMID]
- ↑ (en) Laiko VV, Moyer SC, Cotter RJ, «Atmospheric pressure MALDI/ion trap mass spectrometry», dans Anal. Chem. , vol. 72, no 21, 2000, p. 5239-43 [texte sur PMID]
- ↑ (en) Karas, M. ; Bachmann, D. ; Hillenkamp, F., «Influence of the Wavelength in High-Irradiance Ultraviolet Laser Desorption Mass Spectrometry of Organic Molecules», dans Anal. Chem. , vol. 57, 1985, p. 2935-9
- ↑ (en) Karas, M. ; Bachman, D. ; Bahr, U. ; Hillenkamp, F., «Matrix-Assisted Ultraviolet Laser Desorption of Non-Volatile Compounds», dans Int J Mass Spectrom Ion Proc , vol. 78, 1987, p. 53-68
- ↑ (en) Tanaka, K. ; Waki, H. ; Ido, Y. ; Akita, S. ; Yoshida, Y. ; Yoshida, T., «Protein and Polymer Analyses up to m/z 100 000 by Laser Ionization Time-of flight Mass Spectrometry», dans Rapid Commun Mass Spectrom , vol. 2, no 20, 1988, p. 151-3
- ↑ (en) [pdf] K Markides, Gräslund, A, «Advanced information on the Nobel Prize in Chemistry 2002»
- ↑ (en) Karas M, Hillenkamp F, «Laser desorption ionization of proteins with molecular masses exceeding 10, 000 daltons», dans Anal. Chem. , vol. 60, no 20, 1988, p. 2299-301 [texte sur PMID]
- ↑ (en) Beavis RC, Chait BT, «Matrix-assisted laser-desorption mass spectrometry using 355 nm radiation», dans Rapid Commun. Mass Spectrom. , vol. 3, no 12, 1989, p. 436-9 [texte sur PMID]
- ↑ (en) Karas, M. ; Bahr, U., «Laser Desorption Ionization Mass Spectrometry of Large Biomolecules», dans Trends Anal. Chem. , vol. 9, 1990, p. 321-5
- (en) Cet article est partiellement ou en totalité issu d'une traduction de l'article de Wikipédia en anglais intitulé «Matrix-assisted laser desorption/ionization».
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 12/04/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.




 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité